Introducción
Durante las últimas décadas, hemos podido asistir a la emergencia de la inteligencia artificial (IA) como una fuerza transformadora en múltiples sectores del conocimiento, de los cuales la medicina no podía ser una excepción.
Su capacidad para simular procesos cognitivos humanos, como el aprendizaje, la resolución de problemas y la toma de decisiones, ha abierto nuevas fronteras tanto en la práctica clínica como en la docencia y la investigación1. Dentro del vasto campo de la IA, el aprendizaje automático (machine learning) se ha consolidado como una disciplina fundamental, permitiendo a los sistemas aprender de los datos, sin necesidad de programación explícita, y evolucionar en función de la nueva información disponible. Sin embargo, los mayores avances actuales provienen del campo del aprendizaje profundo (deep learning), gracias al creciente desarrollo de las redes neuronales artificiales.
Estos modelos computacionales están revolucionando la manera en que abordamos problemas complejos: desde el reconocimiento de patrones en grandes volúmenes de datos hasta la predicción de resultados clínicos, pasando por la automatización de tareas diagnósticas y la personalización de tratamientos.
Sin embargo, para que estas herramientas se integren con éxito en la práctica basada en la evidencia, los profesionales sanitarios deben comprender no solo su potencial, sino también sus limitaciones: la necesidad de grandes conjuntos de datos representativos, los riesgos de sesgos y la importancia de la validación externa y la interpretabilidad de los modelos, sin olvidar los aspectos éticos y regulatorios, como la protección de datos, que adquieren especial relevancia en el ámbito pediátrico.
El presente artículo tiene como objetivo explorar los fundamentos de las redes neuronales artificiales, su evolución histórica, su estructura y funcionamiento, los principales tipos de arquitecturas existentes y sus aplicaciones emergentes, con un enfoque particular en la atención sanitaria pediátrica.
¿Qué es una red neuronal artificial?
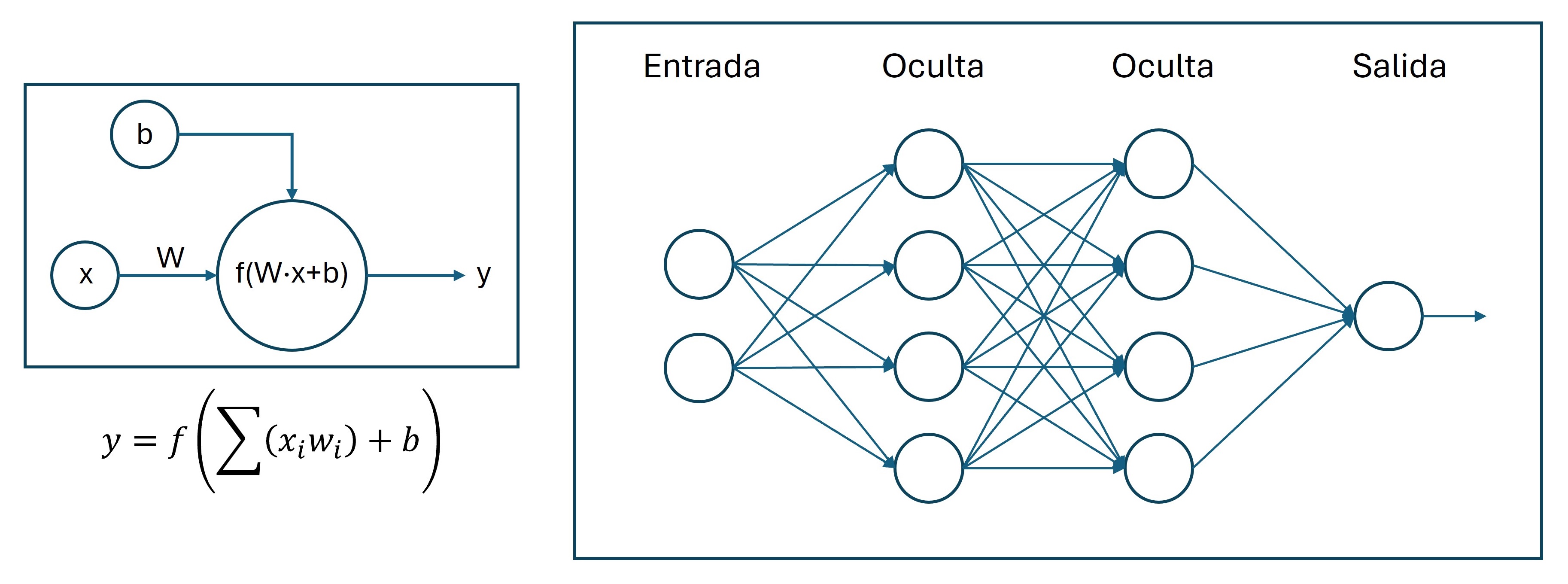
Una red neuronal artificial (RNA) es un modelo computacional inspirado en la organización jerárquica y el mecanismo de transmisión sináptica de las neuronas biológicas. En su forma más básica, una RNA está compuesta por una serie de nodos, llamados neuronas artificiales, distribuidos en capas (entrada, una o varias ocultas y salida)2.
Las RNA están diseñadas para aprender a partir de datos mediante la identificación de patrones complejos y relaciones subyacentes, incluso cuando estas no son evidentes para el análisis humano tradicional.
Una breve perspectiva histórica
Aunque la aspiración de construir “máquinas pensantes” se remonta a los tiempos de la antigua Grecia, los cimientos formales de las RNA se asientan a mediados del siglo XX, cuando Warren McCulloch y Walter Pitts publican el primer modelo matemático de neurona artificial, estableciendo la base teórica sobre la que se edificarán las RNA modernas3,4.
Para ver la primera aplicación real, hay que esperar hasta 1957, momento en que Frank Rosenblatt presenta el perceptrón, una red monocapa capaz de aprender, pero con la limitación de que solo podía realizar clasificación con datos linealmente separables.
Estas limitaciones ralentizan el desarrollo de las RNA hasta las décadas de los 80 y 90 del siglo pasado, en que se produce el renacimiento de las redes neuronales de la mano de la incorporación de capas ocultas, que dio lugar al perceptrón multicapa, capaz de resolver problemas no lineales, y, sobre todo, al impulso definitivo que proporcionó el algoritmo de retropropagación del error (Hinton, Rumelhart y Williams, 1986). No obstante, la limitada potencia de cálculo y la escasez de datos de entrenamiento frenaron su expansión masiva.
Y así llegamos hasta el presente siglo, en el que el aumento exponencial de la capacidad de cómputo (GPU, TPU), la disponibilidad de grandes conjuntos de datos y la aparición de arquitecturas más sofisticadas han impulsado el aprendizaje profundo. Sobresalen las redes recurrentes y sus variantes LSTM (2007) para secuencias temporales, las redes convolucionales (relanzadas en 2012), y, más recientemente, las redes transformadoras (transformers) (2017), que han revolucionado el procesamiento del lenguaje natural5. Finalmente, la explosión de estas nuevas tecnologías se acelera de manera exponencial con el lanzamiento de ChatGPT el 30 de noviembre de 20226.
Estructura y fundamento de una red neuronal artificial
Una RNA se organiza típicamente en capas de neuronas (Figura 1). La primera capa, denominada capa de entrada, recibe los datos iniciales del problema a analizar. Cada neurona en esta capa representa una variable de entrada. A continuación, se disponen una serie de capas ocultas o profundas (de ahí el nombre de aprendizaje profundo), donde ocurren la mayoría de los cálculos y transformaciones de los datos. La última capa, llamada capa de salida, es la que produce el resultado final de la red. El número de neuronas de esta capa dependerá del tipo de problema analizado (por ejemplo, una neurona para regresión, dos para clasificación binaria y múltiples neuronas para clasificación multiclase)2.
Figura 1. Estructura básica simplificada de una red neuronal artificial. x: entrada; W: peso; b: sesgo; f: función de activación; y: salida. Mostrar/ocultar
Cada neurona individual recibe los resultados de todas las neuronas de la capa anterior (excepto las de la capa de entrada, que recibe una variable cada neurona). Todas estas entradas son ponderadas multiplicándolas por un parámetro denominado peso, se les añade otro parámetro denominado sesgo y, al resultado, se le aplica una función no lineal, llamada función de activación. Se produce así el resultado del procesamiento de la neurona, que se pasa a las neuronas de la siguiente capa (excepto en la capa de salida, en la que constituye el resultado de la red) (Figura 1).
Este sencillo esquema de multiplicación, suma y no-linealidad permite a la red aproximar funciones altamente complejas, dotándola de una capacidad de representación que supera con creces el análisis estadístico clásico cuando se trata de datos de gran dimensionalidad. Para ello, el algoritmo deberá “aprender” el valor óptimo de los pesos y los sesgos, que son los llamados parámetros de la red.
¿Cómo aprenden las redes neuronales? El proceso de entrenamiento
El entrenamiento de una RNA consiste en ajustar iterativamente sus pesos y sesgos para que aprenda a resolver una tarea concreta a partir de un conjunto de datos2. Tras una inicialización aleatoria de los parámetros, cada ejemplo de entrada se propaga hacia adelante por las capas de la red hasta generar una predicción (Figura 2).
Figura 2. Esquema de entrenamiento de una red neuronal artificial. Mostrar/ocultar
En un caso típico de aprendizaje automático supervisado, esa salida se compara con el valor o etiqueta real mediante una función de pérdida o coste, que cuantifica el error. Como es lógico, al haberse inicializado los parámetros de forma aleatoria, la predicción se parecerá poco al valor real de los datos.
A continuación, el algoritmo de retropropagación calcula la contribución al error de cada parámetro de la red y, con ayuda de otro algoritmo (el optimizador), corrige los pesos en sentido opuesto al gradiente de error, escalándolos en función de una cantidad que determina el investigador y que se denomina tasa de aprendizaje.
Este ciclo (paso hacia delante, cálculo del error, retropropagación y actualización) se repite durante múltiples iteraciones o épocas, hasta que la red alcanza el rendimiento deseado o se cumple un criterio de parada preestablecido. A lo largo de cientos o miles de ciclos, la RNA va descubriendo patrones latentes y relaciones no evidentes, lo que la habilita para ejecutar tareas de clasificación, regresión, agrupación o generación de datos.
Además de los parámetros, existen hiperparámetros que no se aprenden directamente de los datos, sino que se configuran antes del entrenamiento. Estos incluyen la tasa de aprendizaje, el número de capas ocultas, el número de neuronas por capa, la función de activación, la función de coste, el número de épocas y técnicas de regularización, entre otros. La elección adecuada de los hiperparámetros es esencial para el buen rendimiento del modelo tras la fase de entrenamiento.
Principales arquitecturas de redes neuronales
La estructura descrita hasta el momento corresponde a la de la red neuronal densa o perceptrón multicapa. Esta es la arquitectura básica donde cada neurona en una capa está completamente conectada a cada neurona de la siguiente capa. Estas redes se utilizan comúnmente en tareas sencillas de clasificación y regresión.
Existen, además, diversas arquitecturas de RNA, cada una adaptada a diferentes tipos de datos y tareas2:
- Redes neuronales recurrentes: diseñadas para manejar datos secuenciales o temporales (por ej.: texto, series de tiempo), donde el orden es importante. Las neuronas tienen conexiones que forman ciclos, permitiendo que la información de pasos anteriores persista, lo que las dota de memoria, aunque limitada. Existen variantes como las redes LSTM (Long Short-Term Memory) y GRU (Gated Recurrent Unit), que son efectivas para modelar dependencias a más largo plazo y pueden usarse en el procesamiento de lenguaje natural (PLN), aunque, en la actualidad, se ven superadas por otras arquitecturas más complejas.
- Redes neuronales convolucionales: especializadas en el procesamiento de datos con estructura de rejilla, como imágenes y video, a los que aplican una función especializada denominada convolución. Son la base de muchos avances en visión por computadora y diagnóstico por imágenes en medicina.
- Autocodificadores (autoencoders): redes diseñadas para el aprendizaje no supervisado, capaces de aprender representaciones compactas de los datos. Constan de un codificador, que comprime la entrada hasta formar una nueva representación de los datos, en el llamado “cuello de botella”, y un decodificador, que intenta reconstruir la entrada original a partir de la representación codificada. Son útiles para compresión de datos, eliminación de ruido y detección de anomalías. Una variante especializada para segmentación de imágenes es la U-Net.
- Redes generativas antagónicas: compuestas por dos sub-redes, un generador y un discriminador, que “compiten” entre sí. El generador intenta crear datos sintéticos realistas (por ej.: imágenes), mientras que el discriminador intenta distinguir entre datos reales y los generados. Este proceso lleva a la generación de datos de alta calidad y se usa en generación de imágenes, videos y datos sintéticos, incluso para mejorar imágenes médicas.
- Redes atencionales y transformadoras (transformers): arquitecturas más recientes basadas en mecanismos de “atención”, que permiten al modelo ponderar la importancia de diferentes partes de los datos de entrada. La “auto-atención” (self-attention) les permite procesar secuencias de manera paralela, mejorando la eficiencia respecto a las redes neuronales recurrentes. Han revolucionado el PLN (traducción automática, generación de texto) y se aplican también a imágenes, video y audio. Son la base de los modelos grandes de lenguaje (LLM).
- Modelos grandes de lenguaje: son extensiones masivas de los modelos transformadores, entrenados con enormes cantidades de datos y miles de millones de parámetros. Demuestran capacidades avanzadas en comprensión y generación de lenguaje, razonamiento y resolución de problemas en múltiples dominios. Son la base de aplicaciones conversacionales como ChatGPT.
Aplicaciones de las redes neuronales en medicina
El potencial de las RNA para transformar la medicina es inmenso, abarcando desde el diagnóstico hasta el desarrollo de tratamientos y la gestión hospitalaria. Algunas áreas destacadas incluyen:
- Diagnóstico por imagen: las redes convolucionales son particularmente efectivas en el análisis de imágenes médicas (radiografías7,8, TAC, resonancias magnéticas9, histopatología10) para la detección temprana y precisa de enfermedades como neumonía11, fracturas, tumores12, retinopatía diabética y cáncer de mama. Pueden identificar patrones sutiles que escapan al ojo humano y agilizar el proceso diagnóstico13.
- Pediatría y Neonatología: en el ámbito pediátrico, donde el diagnóstico puede ser complejo, las RNA ayudan en la detección de enfermedades congénitas, el control de signos vitales en unidades de cuidados intensivos neonatales y la predicción de riesgos, como el parto prematuro.
- PLN en medicina: los LLM permiten analizar grandes volúmenes de texto médico (historias clínicas, artículos científicos) para extraer información relevante, ayudar en la recomendación de tratamientos y desarrollar asistentes virtuales para la atención primaria o la automatización de tareas administrativas14,15.
- Genómica y medicina de precisión: las RNA (redes densas, transformadoras) analizan datos genómicos y “ómicos”16,17 para identificar biomarcadores de enfermedades, predecir el riesgo genético individual y personalizar tratamientos, optimizando la eficacia y minimizando efectos adversos18.
- Predicción de enfermedades crónicas: modelos basados en redes densas y recurrentes pueden predecir la evolución y complicaciones de enfermedades crónicas, como diabetes, insuficiencia renal, asma o patologías cardiovasculares, permitiendo intervenciones preventivas19,20.
- Optimización de procesos hospitalarios: se utilizan para mejorar la gestión de recursos, optimizar el flujo de pacientes, predecir la estancia hospitalaria, gestionar los tiempos de espera y realizar triajes automatizados en urgencias15.
- Farmacología y desarrollo de medicamentos: las redes recurrentes y las antagónicas contribuyen al descubrimiento de nuevos fármacos (por ej.: AlphaFold para estructuras proteicas21), la optimización de dosis y la predicción de interacciones y efectos adversos.
- Medicina física y rehabilitación: se emplean en el desarrollo de prótesis inteligentes, el seguimiento de programas de rehabilitación mediante sensores y el análisis de patrones de recuperación, utilizando redes densas y recurrentes.
- Telemedicina: facilitan el diagnóstico remoto asistido por IA, la monitorización continua de pacientes crónicos en sus hogares y la generación de alertas en tiempo real22,23.
- Salud Pública y Epidemiología: las RNA ayudan a modelar la propagación de enfermedades, detectar tempranamente brotes epidémicos y analizar determinantes sociales de la salud para informar políticas preventivas24.
Retos y futuro de las redes neuronales en medicina
Las RNA protagonizan ya avances notables en investigación y práctica clínica, pero su adopción generalizada sigue enfrentando retos importantes.
Para que un modelo sea fiable, necesita entrenarse con grandes volúmenes de datos rigurosamente etiquetados y representativos; de lo contrario, los sesgos estadísticos pueden afianzar, o incluso ampliar, las inequidades sanitarias existentes25,26. Además, la naturaleza opaca de muchos algoritmos (“cajas negras”) dificulta comprender y auditar el razonamiento que conduce a una recomendación diagnóstica o terapéutica, un requisito ético y legal en entornos críticos como la Pediatría. A ello se suman la protección de la privacidad y la seguridad de la información sensible de los pacientes, que exigen marcos sólidos de gobernanza de datos, y la necesidad de validar y regular estas herramientas con el mismo rigor que cualquier dispositivo médico antes de su uso asistencial.
Pese a estos desafíos, el horizonte es alentador27: la investigación avanza hacia modelos más interpretables, transparentes y robustos, capaces de integrar simultáneamente imágenes, texto clínico, información genómica y datos procedentes de dispositivos portátiles para ofrecer decisiones cada vez más precisas y personalizadas. En definitiva, las RNA han pasado de ser un concepto teórico a convertirse en una tecnología revolucionaria que ya redefine el diagnóstico, el tratamiento y la investigación biomédica. Superar los retos descritos será clave para que su impacto positivo se extienda a toda la práctica clínica basada en la evidencia.
Bibliografía
- Weissler EH, Naumann T, Andersson T, Ranganath R, Elemento O, Luo Y, et al. The role of machine learning in clinical research: transforming the future of evidence generation. Trials. 2021;22:537.
- Chollet F, Kalinowski WT, Allaire JJ. Deep learning with R. 2º Ed. New York: Manning Publications Co; 2022.
- Breve historia visual de la inteligencia artificial. En: National Geographic España [en línea] [consultado el 18/06/2025]. Disponible en www.nationalgeographic.com.es/ciencia/breve-historia-visual-inteligencia-artificial_14419?utm_medium=picks.es.20200917&utm_source%C3%A2%C2%80%C2%A6
- Negnevitsky M. The history of artificial intelligence or from the 'Dark Ages' to the knowledge-based systems. WIT Transactions on Information and Communication Technologies; 1997 [en línea] [consultado el 18/06/2025]. Disponible en www.witpress.com/elibrary/wit-transactions-on-information-and-communication-technologies/19/13860
- Vaswani A, Shazeer N, Parmar N, Uszkoreit J, Jones l, Gomez AN, et al. Attention is all you need. 31st Conference on Neural Information Processing Systems (NIPS 2017). Long Beach, CA, USA [en línea] [consultado el 18/06/2025]. Disponible en https://proceedings.neurips.cc/paper/2017/file/3f5ee243547dee91fbd053c1c4a845aa-Paper.pdf
- Introducing ChatGPT. En: OpenAI [en línea] [consultado el 18/06/2025]. Disponible en https://openai.com/index/chatgpt/
- Larson DB, Chen MC, Lungren MP, Halabi SS, Stence NV, Langlotz CP. Performance of a deep-learning neural network model in assessing skeletal maturity on pediatric hand radiographs. Radiology. 2018;287:313-22.
- Summers RM. Deep learning lends a hand to pediatric radiology. Radiology. 2018;287:323-5.
- Hazlett HC, Gu H, Munsell BC, Kim SH, Styner M, Wolff JJ, et al. Early brain development in infants at high risk for autism spectrum disorder. Nature. 2017;542:348-51.
- Ahmad Z, Rahim S, Zubair M, Abdul-Ghafar J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diag Pathol. 2021;16:24.
- Saboo YS, Kapse S, Prasanna P. Convolutional neural networks (CNNs) for pneumonia classification on pediatric chest radiographs. Cureus. 2023;15:e44130.
- Bhinder B, Gilvary C, Madhukar NS, Elemento O. Artificial intelligence in cancer research and precision medicine. Cancer Discov. 2021;11: 900-15.
- Lejeune A, Le Glaz A, Perron PA, Sebti J, Baca-Garcia E, Walter M, et al. Artificial intelligence and suicide prevention: a systematic review. Eur Psychiatry. 2022;65:1-22.
- Kim H, Jin HM, Jung YB, You SC. Patient-friendly discharge summaries in korea based on ChatGPT: software development and validation. J Korean Med Sci. 2024;39:e148.
- Ayers JW, Poliak A, Dredze M, Leas EC, Zhu Z, Kelley JB, et al. Comparing physician and artificial intelligence chatbot responses to patient questions posted to a public social media forum. JAMA Intern Med. 2023;183:589-96.
- Wu J, Singleton SS, Bhuiyan U, Krammer L, Mazumder R. Multi-omics approaches to studying gastrointestinal microbiome in the context of precision medicine and machine learning. Front Mol Biosci. 2023;10:1337373.
- Sha Y, Meng W, Luo G, Zhai X, Tong HHY, Wang Y, et al. MetDIT: transforming and analyzing clinical metabolomics data with convolutional neural networks. Ann Chem. 2024.
- Javaid A, Shahab O, Adorno W, Fernandes P, May E, Syed S. Machine learning predictive outcomes modeling in inflammatory bowel diseases. Inflamm Bowel Dis. 2022;28:819-29.
- Katzman JL, Shaham U, Cloninger A, Bates J, Jiang T, Kluger Y. DeepSurv: personalized treatment recommender system using a Cox proportional hazards deep neural network. BMC Med Res Methodol. 2018;218:24.
- Yang B, Liu C, Wu R, Zhong J, Li A, Ma L, et al. Development and validation of a DeepSurv nomogram to predict survival outcomes and guide personalized adjuvant chemotherapy in non-small cell lung cancer. Front Oncol. 2022; 12:895014.
- NovaFold AI Powered by AlphaFold 2. En: DNA STAR [en línea] [consultado el 18/06/2025]. Disponible en www.dnastar.com/software/nova-protein-modeling/novafold-ai-alphafold-2/?gclid=CjwKCAjw_MqgBhAGEiwAnYOAelDvo7qfHlCBJB0Z6hFPCDmTSQ_MVe7wC_lNL-gxi5UbFa1aq96WBxoCzLAQAvD_BwE
- Hughes A, Shandhi MH, Master H, Dunn J, Brittain E. Wearable devices in cardiovascular medicine. Circ Res. 2023;132:652-70.
- Shumba AT, Montanaro T, Sergi I, Bramanti A, Ciccarelli M, Rispoli A, et al. Wearable technologies and ai at the far edge for chronic heart failure prevention and management: a systematic review and prospects. Sensors. 2023;23:6896.
- Lezcano A, Llamosas G, López A, Parra FJ. Un análisis de modelos de pronóstico para COVID-19 en Cantabria. Instituto Cántabro de Estadística [en línea] [consultado el 18/06/2025]. Disponible en https://jecas.es/wp-content/uploads/2021/11/13.2.ICANE_COVID.pdf
- Uche-Anya E, Anyane-Yeboa A, Berzin TM, Ghassemi M, May FP. Artificial intelligence in gastroenterology and hepatology: how to advance clinical practice while ensuring health equity. Gut. 2022;71:1909-15.
- Istasy P, Lee WS, Iansavichene A, Upshur R, Gyawali B, Burkell J, et al. The impact of artificial intelligence on health equity in oncology: scoping review. J Med Internet Res. 2022;24:e39748.
- Mondillo G, Perrotta A, Colosimo S, Frattolillo V, Masino M, Pettoello-Mantovani M. Artificial intelligence in pediatrics: an opportunity to lead, not to follow. J Pediatr. 2025.
Cómo citar este artículo
Molina Arias M. Redes neuronales artificiales: fundamentos y aplicaciones. Evid Pediatr. 2025;21:25.

Envío de comentarios a los autores